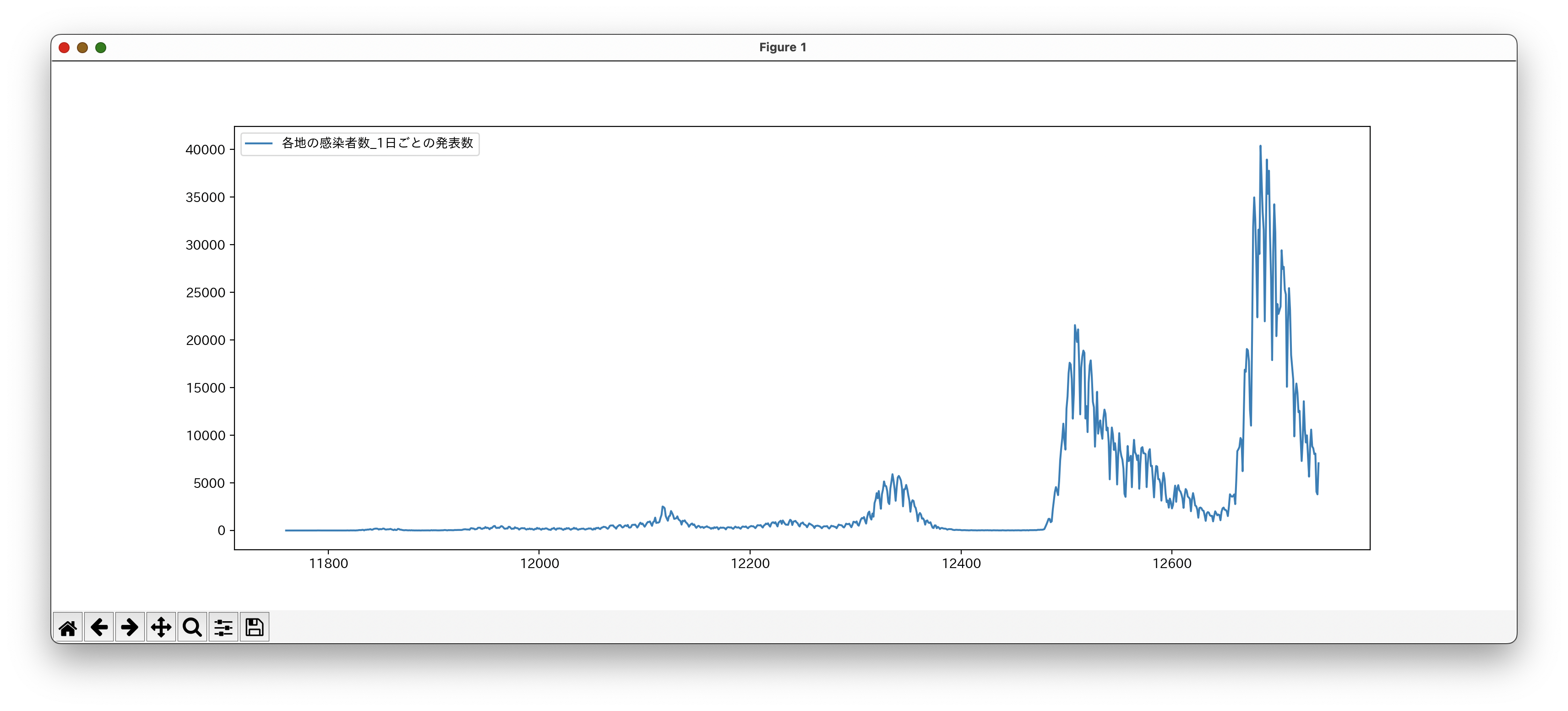
第一波からの推移をスクロールしないで見たい
かれこれ2年以上新型コロナウイルスの流行が続いています。
やっぱりこれだけ長く続くと疲れるというか飽きるというか、いろいろ面倒くさくなってくるわけですが、それじゃまずいので、現在の感染状況だけは把握するようにしています。
毎日見るのはNHKのコロナサイトです。
都道府県別の感染者数や死者数のグラフがすぐに見られて便利です。
ただ、グラフの一部しか見られないので長期的なトレンドが分かりずらいです。
ありがたいことにCSVデータがダウンロードできるので、それを使って自分でグラフを描いてみることにしました。
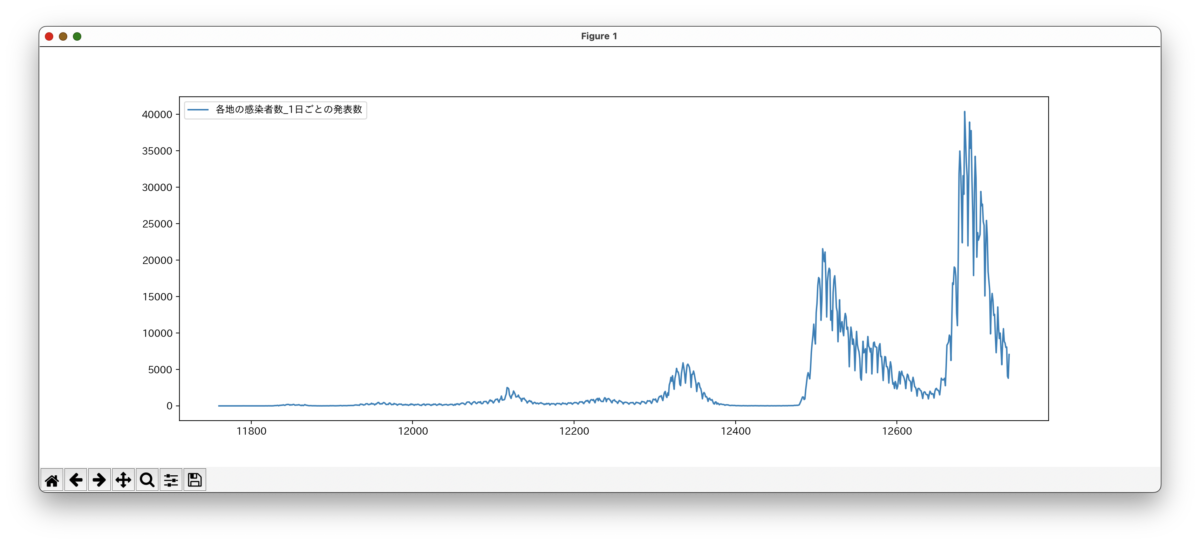
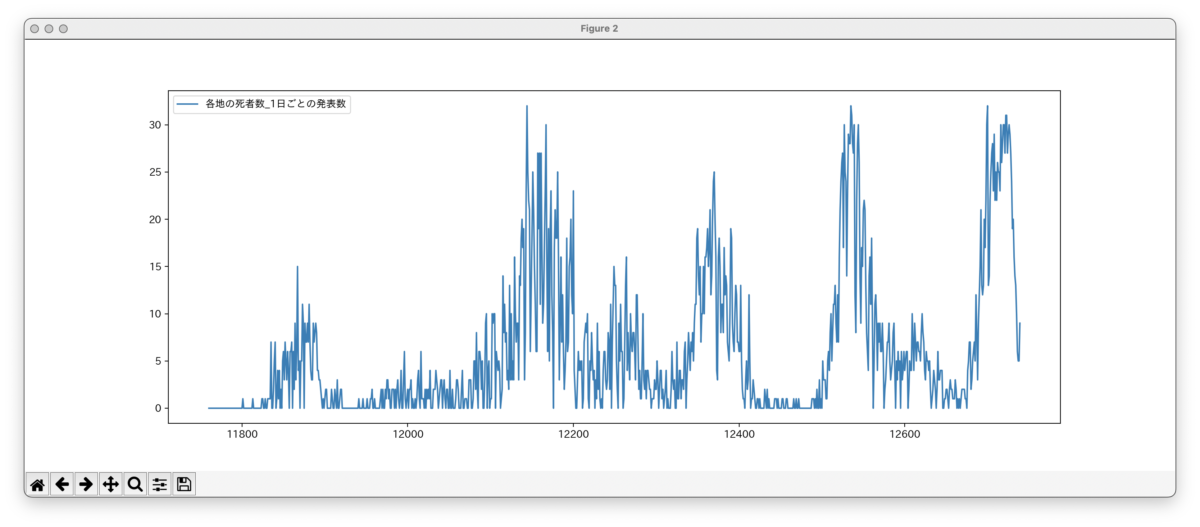
とりあえず東京都の感染者数と死者数を毎日グラフで表示して自分なりに状況を把握しています。
コード
言語はPythonで、ライブラリとしてはpandasとmatplotlibを使っています。
とりあえずコードは以下のような感じです。
ライブラリのインストール
pip install pandas
pip install matplotlib
pip install japanize-matplotlib
// Jupyter Labの場合は先頭に"!"を付ける
!pip install pandas
!pip install matplotlib
!pip install japanize-matplotlib
Pythonコード
import pandas as pd
import matplotlib
import matplotlib.pyplot as plt
import japanize_matplotlib
matplotlib.pyplot.rcParams['figure.figsize'] = (16.0, 6.0) #グラフのサイズ設定
data = pd.read_csv(
'https://www3.nhk.or.jp/n-data/opendata/coronavirus/nhk_news_covid19_prefectures_daily_data.csv')
tokyo = data.query('都道府県名=="東京都"')[
["日付", "各地の感染者数_1日ごとの発表数", "各地の死者数_1日ごとの発表数"]]
tokyo[["日付", "各地の感染者数_1日ごとの発表数"]].plot()
tokyo[["日付", "各地の死者数_1日ごとの発表数"]].plot()
plt.show() #Jupyter Lab上では不要
例えば、corona-plot.py という名前でコードを保存してあれば、ターミナルで
python3 corona-plot.py とやれば少ししてグラフが描かれます。
Jupyter Lab上で実行するばあいにはplt.show()がなくても自動的にグラフが表示されます。
なぜか日付が数字になってしまいます。直す方法を調べたのですが、ちょっと面倒くさそうなので放置しています。トレンドを見る分には問題ないです。
pandasとmatplotlibの基本
今回はpandasとmatplotlibの基本的なコマンドだけを使いました。
使い方を簡単にメモしておきます。
データフレームへの読み込み
まず、CSVデータをpandasのデータフレームに読み込んで、いろんな処理ができるようにします。
import pandas as pd
data = pd.read_csv('{url もしくは path}')
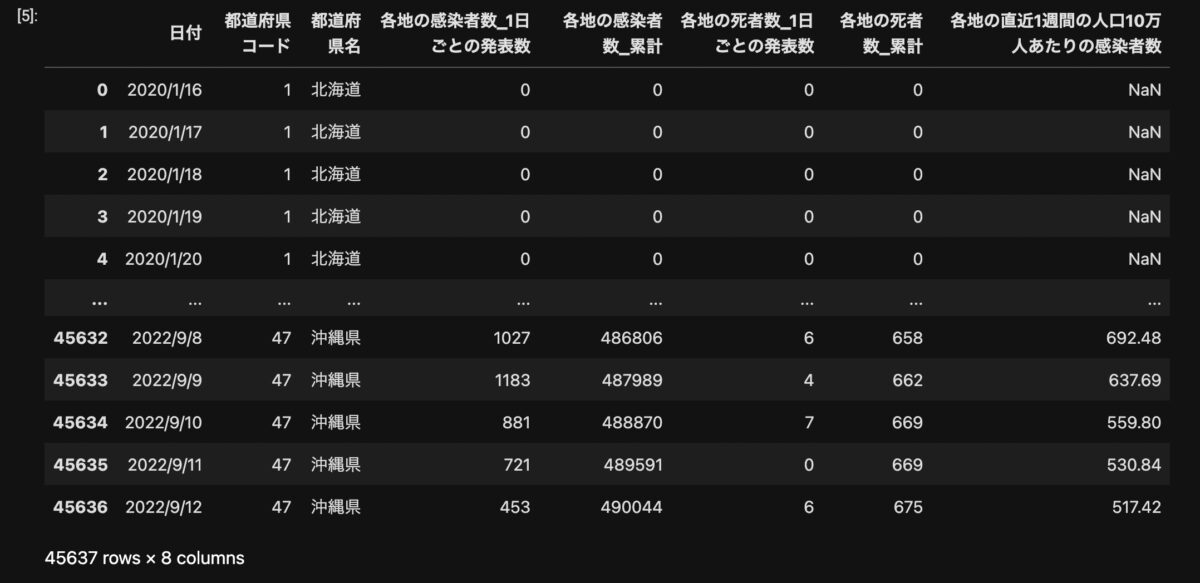
条件によるデータの抽出
data.query()で条件を設定してデータを絞り込みできます。
tokyo = data.query('都道府県名=="東京都"')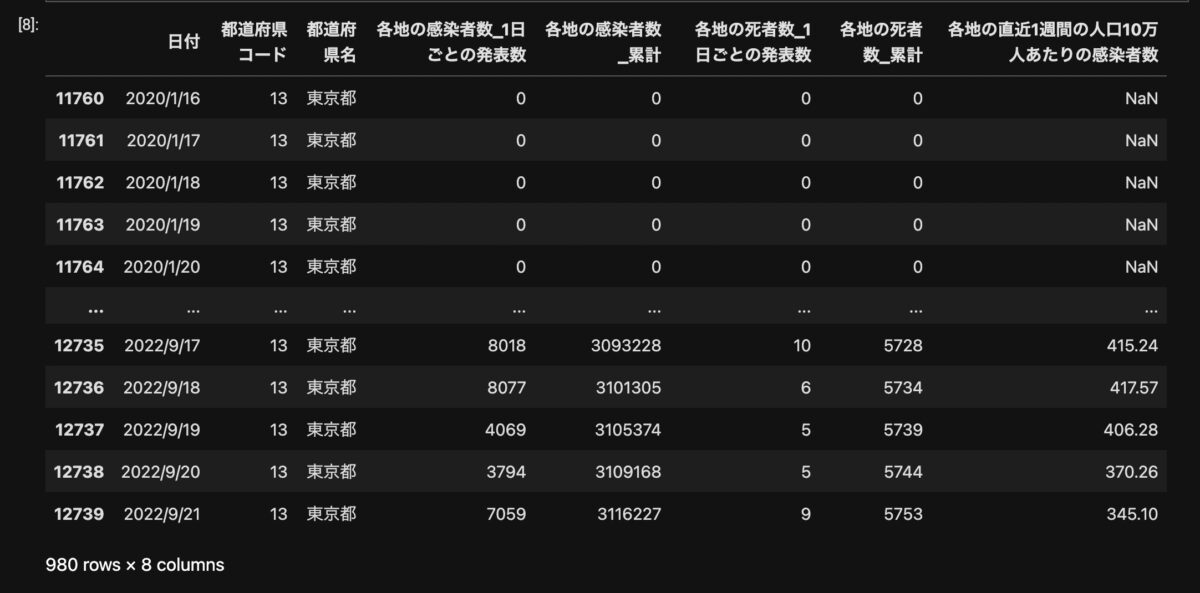
任意の項目だけを抜き出し
表の中から特定の項目だけを抜き出します。
selected = tokyo[["日付", "各地の感染者数_1日ごとの発表数", "各地の死者数_1日ごとの発表数"]]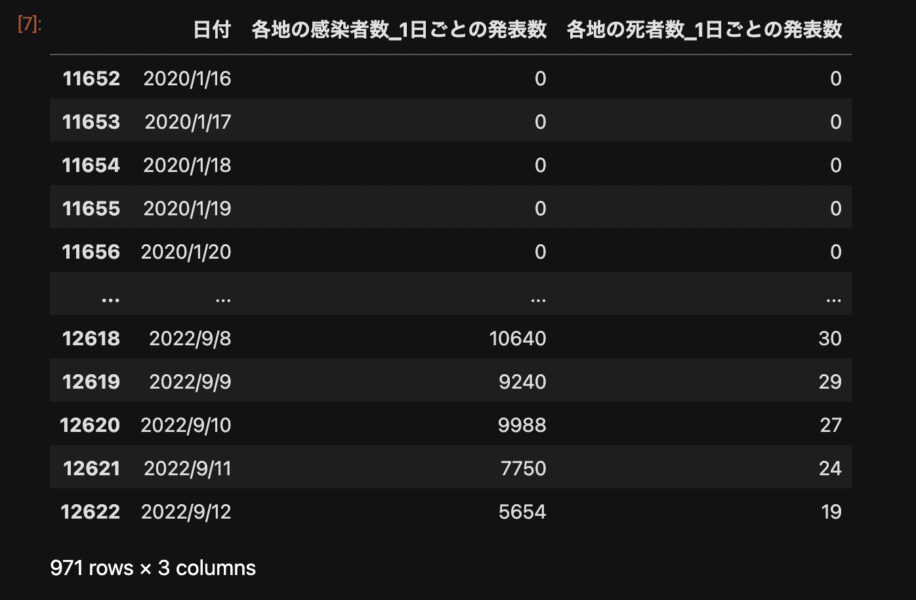
グラフのプロット
グラフにしたい項目をリストで指定して.plot()メソッドを呼びます。
デフォルトでは折れ線グラフになるみたいです。
コマンドライン上で実行する場合にはplt.show()で表示させます。
tokyo[["日付", "各地の感染者数_1日ごとの発表数"]].plot()
plt.show()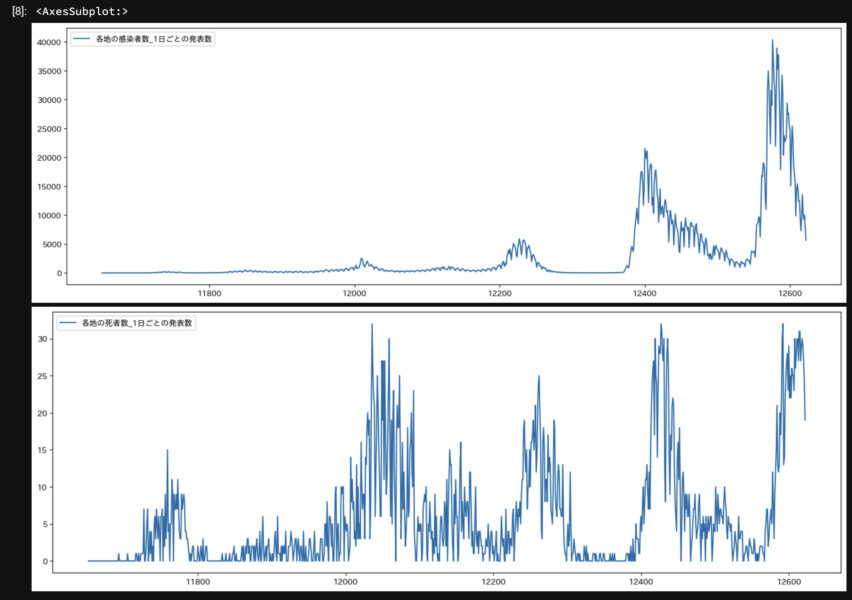
表示の設定とかを凝り出すとキリがないですが、ちょっとトレンドを見たいという時に簡単にグラフが描けて便利ですね。